活動
2021年2月19日
「若手研究者データ解析・共有基盤創出チャレンジ」研究成果報告

神経幹細胞における、分化運命決定因子の
ダイナミックな発現変動の機能的意義の解明
計画研究A02今吉班
京都大学 生命科学研究科 高次生命科学専攻 脳機能発達再生制御学
博士後期課程1年 長崎 真治
次世代シークエンサーを用い、ターゲット細胞 (または細胞群) の遺伝子発現を網羅的に定量解析できるRNA-seqが生命科学の研究で広まっている。私自身、所属研究室で実施されているRNA-seqの研究にはいくつか携わってはいたが、それらは細胞培養やフローサイトメトリーを利用したターゲット細胞の分離など、いわゆる"ウェット"な実験であり、取得したデータをより詳しく解析するためのバイオインフォマティクスや数理統計解析といった"ドライ"な実験にはあまり携わってこなかった。このような状況は、多くの生命科学研究者にも当てはまり、つまり、"ウェット"と"ドライ"の実験の両者を実施できる研究者は多くないと思う。しかし、このようなことが言っていられるのも今のうちだけで、数年後にはRNA-seqに関しては、"ウェット"と"ドライ"の実験を自身一人で実施できないと時代遅れな研究者になってしまうと感じる。(もしくはすでに時代遅れになっているのかもしれないが。)
このようなRNA-seqに基づくデータなしでは語れないようになってきている現状の中で、「若手研究者 データ解析・共有基盤創出チャレンジ」に採択いただき、"ウェット"な実験がメインだった私に、"ドライ"な実験に精通できる場を提供してくださり、RNA-seq取得データに対する解析手法・考察力を拡張することができた。特に、これまでは他人に解析依頼した結果のみから考察するだけであったが、本チャレンジにおいてデータ解析手法を習得したことで、さらに踏み込んだ解析を自身で実施できるようになった。
今後は、本チャレンジを通して特定した神経幹細胞の分化運命決定に関わる新規遺伝子群や、それらの発現パターンの機能的意義の解明を目指し、"ウェット"と"ドライ"の実験を自由自在に行き来し、なおかつ、ありふれたRNA-seqの研究枠に収まらない、オリジナリティのある研究を実施していきたい。
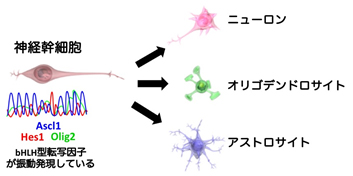
図. 神経幹細胞の分化
